L’acetilazione si riferisce a una reazione che introduce un gruppo funzionale acetile in un composto chimico, in cui l’atomo di idrogeno di un gruppo idrossile viene sostituito da un gruppo acetile (CH3 CO) per produrre un estere specifico, l’acetato. L’acetilazione delle proteine ha comunemente due forme diverse. Negli esseri umani, quasi (80%-90%) le proteine vengono acetilate co-translazionalmente ai loro terminali Nα delle catene polipeptidiche nascenti. Un altro tipo è tipicamente acetilato sui residui di lisina.
Acetilazione N-terminale
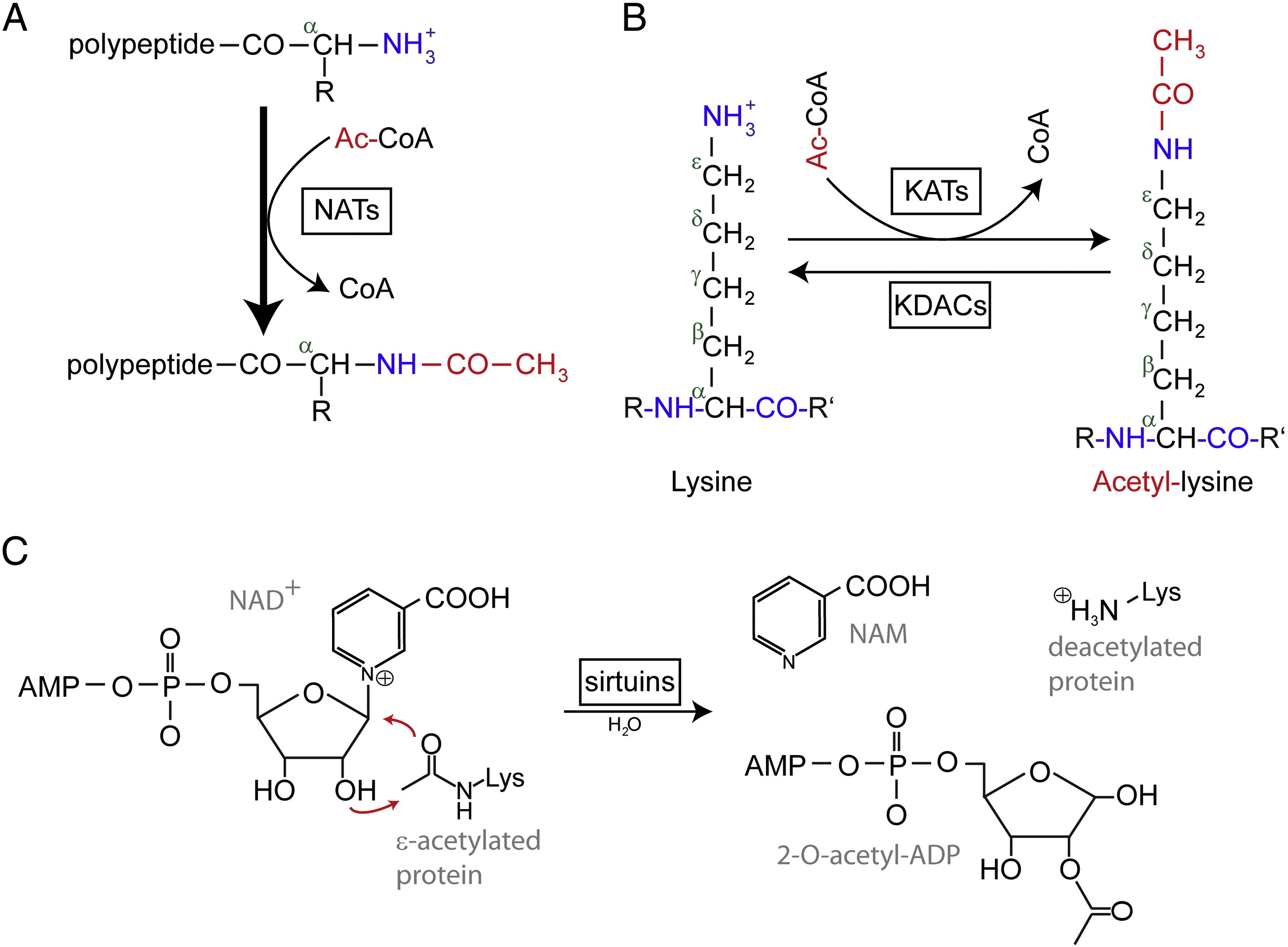
L’acetilazione N-terminale (Nt) è catalizzata da Nt-acetiltransferasi (NATs) ed è trovata essere irreversibile finora. Le NATs, enzimi mono- o multisubunità che consistono in una subunità catalitica e fino a due subunità ausiliarie, possono trasferire un gruppo acetile dall’acetil-coenzima A (Ac-CoA) al gruppo α-ammino del primo residuo aminoacidico della proteina. Nei NAT, la subunità ausiliaria principale modula l’attività e la specificità del substrato della subunità catalitica. Diverse NAT sono responsabili dell’acetilazione dell’Nt. Negli esseri umani sono state trovate finora sei NAT, tra cui NatA, NatB, NatC, NatD, NatE e NatF. Oltre alla differenza nella composizione delle subunità, le varie NAT variano nelle loro specificità di substrato.
L’acetilazione di Nt gioca diversi ruoli negli effetti molecolari. In primo luogo, l’acetilazione Nt determina la localizzazione subcellulare di alcune proteine. Per esempio, Arl3 e Grh1, due proteine associate al Golgi, non possono associarsi all’apparato del Golgi quando manca il gruppo Nt-acetile. In secondo luogo, è stato riportato che l’Nt-acetilazione trattiene le proteine nel citosol e inibisce una migrazione di traslocazione post-traslazionale verso il reticolo endoplasmatico (ER) e la via secretoria. Inoltre, l’acetilazione Nt può alterare le proprietà dell’N-terminale per rendere modulate le interazioni proteina-proteina. È stato dimostrato per diverse proteine che l’affinità con i loro partner di legame è aumentata dopo essere state Nt-acetilate. Per esempio, l’enzima coniugante l’ubiquitina E2 Ubc12 subisce l’Nt-acetilazione da parte di NatC che permette una maggiore affinità verso il suo partner di interazione, l’ubiquitina ligasi E3 Dcn1. Inoltre, l’Nt-acetilazione controlla la qualità e la durata della proteina e regola la stechiometria della proteina attraverso la via dell’N-end rule.
L’acetilazione N-terminale ha molte funzioni in fisiologia. I NAT sono essenziali per il normale sviluppo, lo sviluppo delle ossa e dei vasi sanguigni. L’acetilazione N-terminale può regolare la pressione sanguigna, la localizzazione del proteasoma, l’ormone, così come la struttura e la funzione degli organelli. Nella malattia umana, è legata alle malattie neurodegenerative (come il morbo di Alzheimer, il morbo di Parkinson e la demenza a corpi di Lewy) e il cancro (come il cancro ai polmoni, il cancro al seno, il cancro al colon-retto).
L’acetilazione della lisina
I residui di lisina acetilati sono stati scoperti per la prima volta negli istoni che regolano la trascrizione del gene. Ma l’acetilazione della lisina non è limitata agli istoni. A differenza dell’acetilazione Nt, l’acetilazione della lisina è reversibile. L’acetilazione è catalizzata dalle acetiltransferasi della lisina (KATs) e la deacetilazione dei residui di lisina è catalizzata dalle deacetilasi della lisina (KDACs).
Si riporta che 17-22 geni KATs sono stati identificati nel genoma umano (Il numero esatto di KATs è controverso), che possono essere classificati in tre diverse famiglie, tra cui GCN5 (controllo generale non derepressivo 5) – famiglia di acetiltransferasi correlate (GNAT), la famiglia MYST, e p300/CBP (CREB-binding protein) famiglia. I substrati noti dei complessi KAT non includono solo le proteine istone, ma alcuni diversi fattori di trascrizione, co-regolatori trascrizionali, e alcune proteine di specifiche vie di segnalazione cellulare come p53, β-catenina, NF-κB, MyoD o Rb. E anche alcune molecole di RNA possono essere acetilate. Ci sono quattro diversi tipi di KDAC, tra cui Classe I, II, III e IV. Le classi I, II e IV sono amidoidrolasi Zn2 +-dipendenti, mentre la classe III (chiamata anche sirtuine) usa NAD+ come co-substrato per la sua attività catalitica sono amidoidrolasi Zn2 +-dipendenti.
Gli istoni sono le prime proteine acetilate scoperte. Le molecole degli istoni sono modificate da diversi PTM, tra cui fosforilazione, metilazione e acetilazione. L’acetilazione degli istoni determina l’assemblaggio degli istoni così come il ripiegamento e la compattezza dell’interazione DNA-istone e quindi presenta un interruttore tra struttura cromatinica permissiva e repressiva. Oltre agli istoni, le KAT possono catalizzare le proteine citoscheletriche. Ci sono alcuni nuovi siti di acetilazione identificati in altre proteine, comprese le proteine HMG, c-Myc, recettori di estrogeni e androgeni, E2F/Rb e così via.
Sono stati riportati i ruoli fisiologici dell’acetilazione della lisina. Le proteine istone sono associate a una stretta regolazione di essenzialmente tutti i tipi di processi basati sul DNA come la trascrizione, la replicazione, la ricombinazione, la riparazione, così come la traduzione e la formazione di strutture specializzate della cromatina. Pertanto, l’acetilazione della lisina delle proteine influisce su una serie di vie di segnalazione cellulare, nonché sul metabolismo, sulle risposte allo stress, sull’apoptosi e sul traffico di membrana.
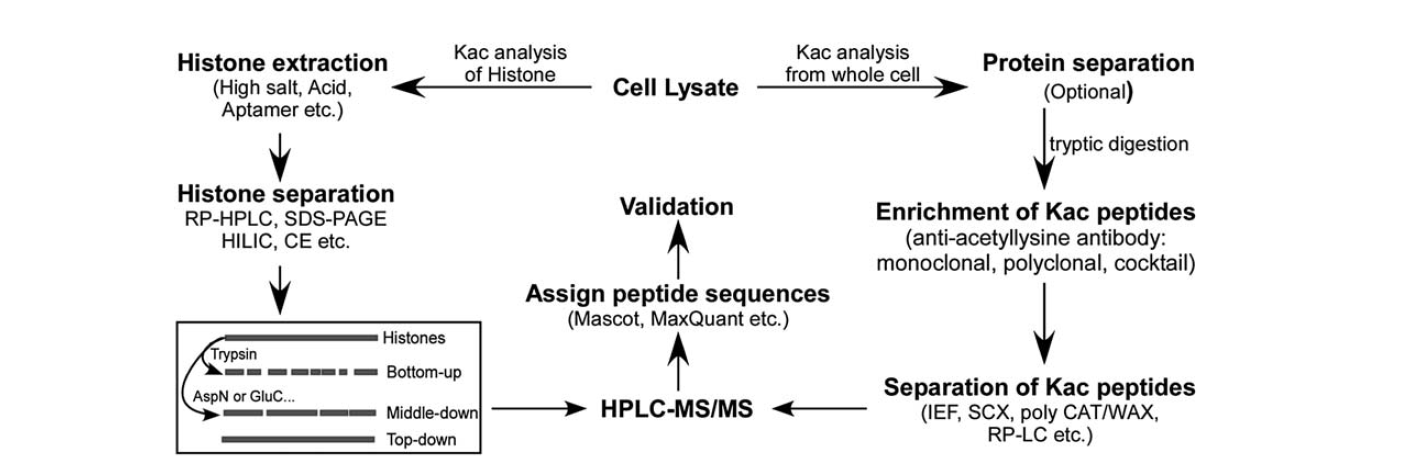
Ci sono relativamente pochi strumenti disponibili per identificare l’acetilazione della lisina. Con lo sviluppo della spettrometria di massa, essa è diventata uno strumento importante per l’identificazione e la quantificazione dell’acetilazione della lisina. Per analizzare l’acetilazione della lisina degli istoni, ci sono diverse fasi, tra cui il lisato cellulare, l’estrazione degli istoni, la separazione degli istoni, la spettrometria di massa e l’analisi dei dati. Le fasi di analisi per l’acetilazione della lisina delle proteine dalla cellula intera includono lisato cellulare, separazione delle proteine, arricchimento dei peptidi di acetilazione della lisina, separazione dei peptidi di acetilazione della lisina, spettrometria di massa e analisi dei dati.